利用第三代单分子测序技术结合光学图谱技术获得真菌接近完整的全基因组序列图谱
mBio 2015 IF:6.975
关键词:光学图谱技术;端粒;DNA 转座元件;第三代单分子测序技术
研究背景
真核生物中存在大量的重复序列,这些重复序列会对基因组组装造成很大的影响。传统的高通量测序技术受制于读长,无法完全解析这些重复序列。第三代单分子测序技术平均读长可达到 10~15 kb,最长读长超过 60 kb,能很好地解析富含重复的区域。光学图谱技术可用于构建高分辨率的图谱。本研究采用第三代单分子测序技术结合光学图谱技术获得了一个 36 Mb 真菌接近完整的全基因组序列图谱。
实验方法
供试样本:棉花黄萎病菌 Verticillium dahliae;
文库大小:22,000 bp,采用 BluePippin 去除 7,000 bp 以下的片段;
测序数据: 14 SMRT Cell P4-C2 + 4 SMRT Cell P5-C3
测序平台:PacBio RS II;
研究结果
(1) 采用单独的 Pacbio 测序数据进行拼接效果最好;
(2) 两步法拼接(第一步采用 A5MiSeq对 MiSeq 数据进行拼接;然后采用PBjelly利用 Pacbio 数据进行拼接)相比一步法(直接采用 SPAdes对MiSeq数据和Pacbio 数据进行拼接)拼接效果更好;
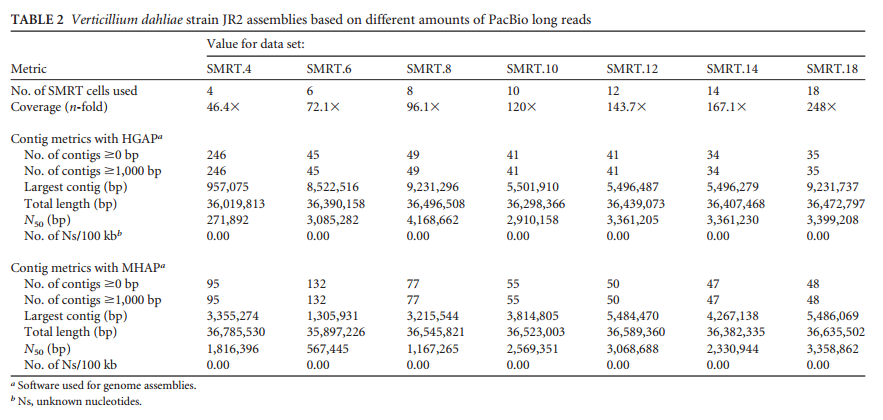
(3)Pacbio 测序数据对拼接效果改善非常显著(见表 1);
(4) 相比 MHAP 软件,HGAP 拼接效果更好(三代测序数据);
(5)当三代测序数据量超过 50 ×,数据量的增加对拼接结果的改善非常显著;当测序数据量超过100 ×,数据量对拼接结果的改善效果不显著 (见表 2)。
参考文献
Luigi Faino , Michael F. Seidl , et al. Single-Molecule Real-Time Sequencing Combined with Optical Mapping Yields Completely Finished Fungal Genome. Mbio, 2014, 6 (4).
项目经验
|
拉丁名 |
基因组大小 |
GC含量 |
派森诺已完成数目 |
|
Saccharomyces pastorianus |
22.3 |
39.25 |
1 |
|
Cerrena.sp |
43.66 |
47.48 |
1 |
|
Chaetomium thermophilum |
28.32 |
52.6 |
1 |
|
Aspergillus terreus |
29.36 |
52.2 |
1 |
|
colletotrichum orbiculare |
90.08 |
37.8 |
1 |
|
cladosporium sphaerospermum |
38.52 |
54.3 |
1 |
|
lentinula edodes |
41.36 |
46 |
1 |
|
Fusarium proliferatum |
44.18 |
48.4 |
2 |
表一
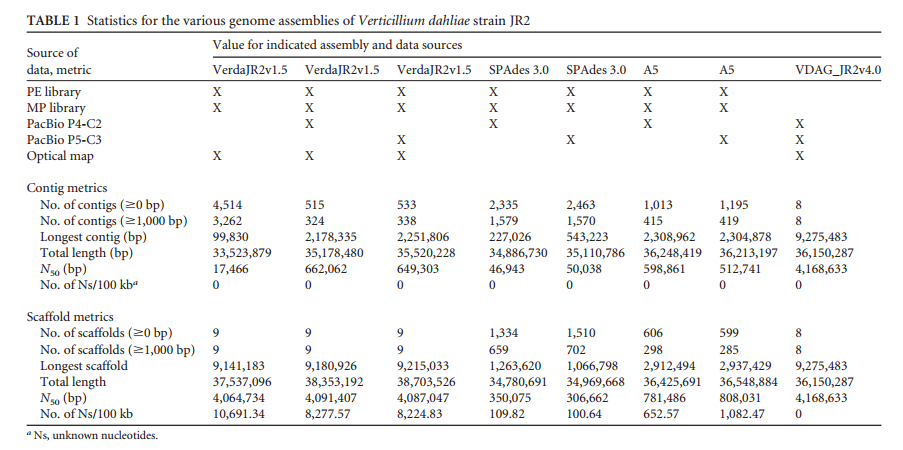
表二